Gli individui con sindrome di Down (DS), la condizione genetica causata dalla trisomia 21, mostrano una forte variabilità interindividuale in termini di fenotipi di sviluppo e diagnosi di condizioni concomitanti. Ricordiamo la crescita stentata, la dismorfogenesi degli organi, o ritardi dello sviluppo neurologico e differenze cognitive, oltre ad un aumento del rischio per una serie di condizioni mediche concomitanti nel corso della vita, tra cui difetti cardiaci congeniti, disturbi dello spettro autistico, disturbi convulsivi, problemi di udito e vista, problemi gastrointestinali, molteplici condizioni autoimmuni, leucemie e morbo di Alzheimer.
Ma nonostante i numerosi progressi nella sua gestione clinica hanno drasticamente esteso l’aspettativa di vita e migliorato la qualità della vita in questa popolazione, la mancanza di comprensione meccanicistica dei fattori che influenzano gli impatti eterogenei sullo sviluppo e clinici di T21 impedisce lo sviluppo di approcci di medicina personalizzata per ulteriori avvantaggiare le persone con DS.
Stabilire relazioni causa-effetto tra la sovraespressione dei geni HSA21 e lo spettro dei tratti associati alla DS si è rivelato difficile. Finora è stata stabilita nell’uomo la connessione tra la triplicazione del gene della proteina precursore dell'amiloide (APP) . E’ però probabile che i geni codificati su HSA21 abbiano interazioni complesse, con potenziali relazioni sia cooperative che antagoniste, che probabilmente influenzano i risultati fenotipici dei tratti poligenici, motivando la necessità di indagini sui contributi dei diversi geni HSA21, da soli o in combinazione, alla variabilità fenotipica della DS.
E’ questo l’obiettivo di uno studio genetico molto complesso, che ha indagato i modelli di espressione dei geni codificati su HSA21 in centinaia di individui con T21 attraverso l'analisi del trascrittoma del sangue intero, e ha portato all'identificazione di due principali sottoinsiemi di geni co-espressi. Utilizzando analisi di clustering, sono stai identificato tre principali sottotipi molecolari di trisomia 21, basati su modelli di sovraespressione differenziale dei geni del cromosoma 21. Successivamente sono state eseguitr analisi comparative multiomiche tra sottotipi utilizzando trascrittomi di sangue intero, proteomi e metabolomi plasmatici e profili di cellule immunitarie, che hanno portato a rilevare una forte eterogeneità nella disregolazione dei processi fisiopatologici chiave nei tre sottotipi, sottolineata da firme multiomiche differenziali correlate all’infiammazione, all’immunità, alla crescita e proliferazione cellulare e al metabolismo, e inoltre evidenziato modelli distinti di cambiamenti delle cellule immunitarie nei sottotipi.
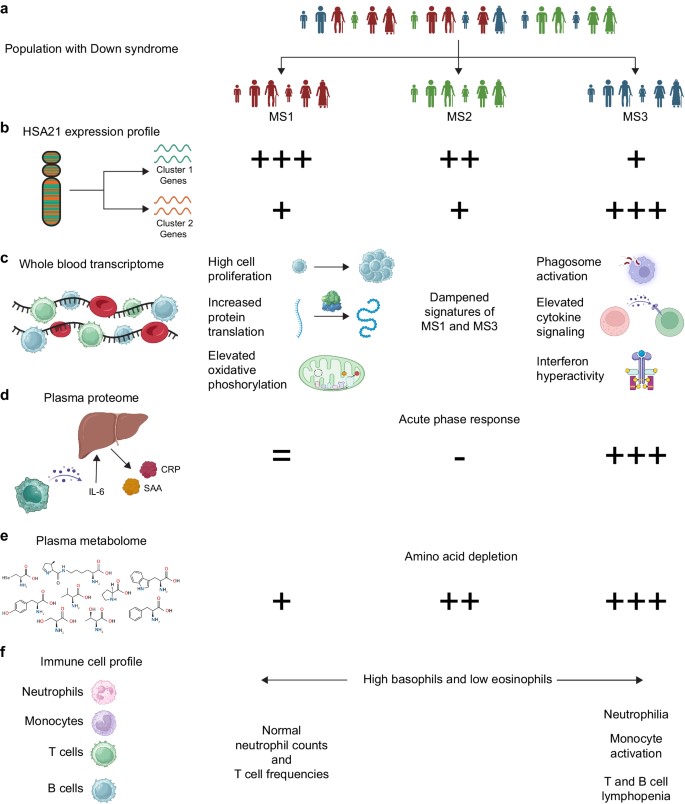
a Gli individui con trisomia 21 (T21) possono essere raggruppati in tre distinti sottotipi molecolari (MS) in base all'espressione dei geni del cromosoma 21 (HSA21). b I geni HSA21 sono espressi in due distinti cluster di co-espressione, il cluster genico HSA21 1 (verde) e 2 (arancione). L'MS1 ha la massima espressione dei geni HSA21 del cluster 1 rispetto ai controlli euploidi (D21, +++), seguito dall'MS2 (++) e dall'MS3 (+). La MS3 presenta la massima espressione dei geni del cluster 2 di HSA21 rispetto alla D21 (+ + +). MS1 e MS2 sovraesprimono i geni del cluster 2 di HS21 a livelli simili (+). c Il trascrittoma del sangue intero di MS1 è caratterizzato da firme di elevata proliferazione cellulare, aumento della traduzione proteica ed elevata fosforilazione ossidativa. La MS3 mostra la più forte upregulation di firme indicative di processi immunitari e infiammatori iperattivi. La MS2 presenta firme attenuate sia nella MS1 che nella MS3. d Rispetto ai controlli euploidi, le firme proteomiche plasmatiche della risposta di fase acuta non sono diverse nella MS1, sono diminuite nella MS2 ed elevate nella MS3. e Rispetto ai controlli euploidi, tutti i sottotipi mostrano una deplezione degli aminoacidi plasmatici, con una gravità crescente dalla SM1 alla SM3. f Tutti i sottotipi mostrano basofili elevati ed eosinofili ridotti, ma solo la SM3 si distingue per una chiara neutrofilia in concomitanza con linfopenia delle cellule T e B.
Questo studio stabilisce che la sovraespressione del gene HSA21 non è presente in modo uniforme nei soggetti con DS, mettendo in discussione la nozione prevalente di sovraespressione coerente di tutti i geni HSA21 nella DS, in quanto rivela che l'entità della sovraespressione varia sia tra i diversi geni HSA21 sia tra gli individui. Questa variabilità, come dimostrato dall'identificazione dei sottotipi molecolari, sembra essere alla base di distinti profili molecolari a valle, suscitando intriganti domande sulla natura della patobiologia della T21. A questo punto non è chiaro se gli MS1-3 rappresentino stati fisiopatologici stabili e distinti o variazioni lungo un continuum. Mentre alcuni dei risultati collocano la MS2 come intermedia lungo uno spettro tra la MS1 e la MS3, essa presenta anche caratteristiche uniche non riscontrate negli altri sottotipi. Questo intricato scenario sottolinea la necessità di un'esplorazione più approfondita, per mettere in relazione queste differenze molecolari con le manifestazioni cliniche della DS, al fine di migliorare la comprensione della diversa patobiologia della DS e di aprire la strada a strategie di cura personalizzate e adattate al panorama molecolare unico degli individui con DS.
Pier Luigi Tucci
Variegated overexpression of chromosome 21 genes reveals molecular and immune subtypes of Down syndrome
Micah G. Donovan, et al.
https://doi.org/10.1038/s41467-024-49781-1
Questi risultati forniscono informazioni sull’eterogeneità molecolare della trisomia 21 e gettano le basi per lo sviluppo di approcci di medicina personalizzata per la gestione clinica della sindrome di Down

